ATAC-seq(Assay for Transposase-Accessible Chromatin with high throughput sequencing)是美国Stanford大学的William Greenleaf教授在2013年所研发的一种全新方法,利用DNA转座酶结合高通量测序技术,来研究染色体的可进入性。ATAC-seq是一种创新的表观遗传学研究技术,该技术通过转座酶对某种特定时空下开放的核染色质区域进行切割,进而获得在该特定时空下基因组中所有活跃转录的调控序列。与其他技术(例如研究相似染色质特征的FAIRE-Seq或DNase-Seq)相比,ATAC-Seq的主要优势在于该测定所需的细胞数量更少,并且其两步法操作相对简单。
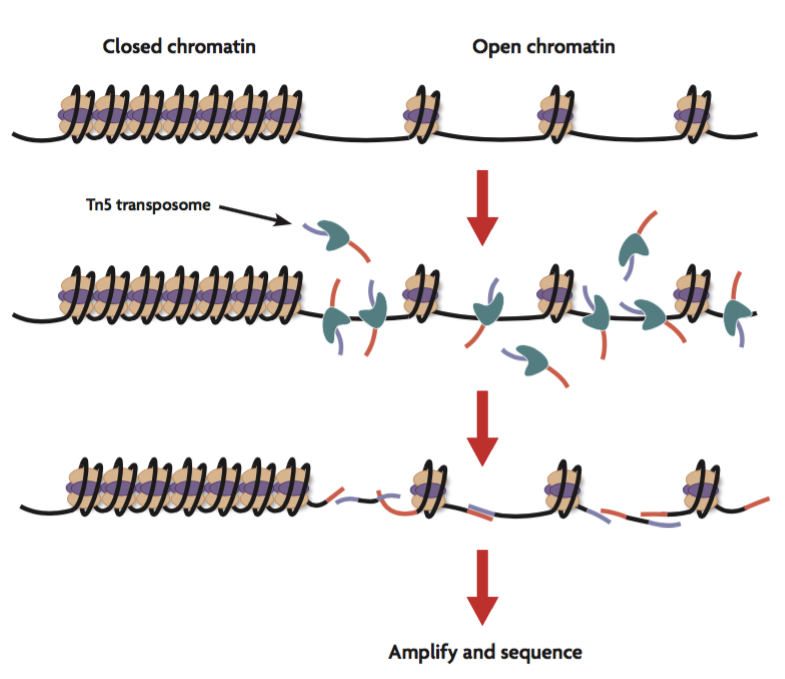
转座子优先并入一般没有核小体(无核小体区域)或暴露DNA段的基因组区域。因此,基因组中某些基因座序列的富集表明该区域不存在核小体,处于DNA结合蛋白等核机器能进入的松散暴露状态,提供有关染色质区段转录活跃状态的信息。
ATAC-seq实验的测序部分通常将产生数百万次可以成功比对到参考基因组的高通量测序reads。每条reads指向在实验期间发生的一次切割事件在基因组上的位置。然后对所比对到基因组上的reads进行计数,并创建具有碱基对分辨率的信号峰值(peak)。在实验过程中DNA可接近的基因组区域(染色质开放区域)含有更多的测序reads(因为这是转座酶优先作用于这些位置)。这些开放位点区域可以进一步分为各种调节元件类型:启动子,增强子,绝缘子等。通过进一步的信息整合,如峰与转录起始位点的距离,产生处于开放区域作用下的基因列表;或计算开放区域内的高倾向结合某种特定蛋白的碱基序列(motif),得到活跃基因的上游调控因子,提供全基因组转录因子发生的信息。
ATAC-seq技术可以进行样本之间差异的染色质开放位点的比较,通常并不单独使用。作为检测表观遗传-染色质开放状态现象的方式,与样本基因表达谱紧密相连,从靶标基因的表观状态关联到表达水平,具有强大的数据挖掘作用。此外,通过关联基因组、外显子,染色质免疫共沉淀(组蛋白修饰或转录因子结合)测序信息,形成:表观调控-基因组-基因表达的完整调控链。
ATAC-Seq服务涵盖实验中的所有步骤,包括膜通透,添加带有接头的转座酶,在开放基因组位置通过转座酶插入接头,文库扩增,Illumina平台上的NGS以及生物信息学分析。我们简要概述了ATAC-Seq实验步骤,该步骤可分为两个主要部分:细胞制备和转座。
细胞制备
第一步需要收获细胞。由于用于ATAC-Seq分析的细胞数量对于转座反应和生成的DNA片段的大小分布至关重要,因此对细胞进行计数非常重要。此外,细胞应完好无损,并且是均匀的单细胞悬浮液。收获后,用非离子型去污剂裂解细胞以产生纯细胞核。
转座反应
然后将所得的染色质片段化,并同时使用Tn5转座酶进行测序接头标记,以生成ATAC-Seq文库。纯化后,可使用barcode引物通过PCR扩增文库。所得文库随后通过qPCR或NGS进行分析。
其他ATAC-Seq注意事项
使用正确的细胞数量用于转座是很重要的。一般建议使用25,000-75,000个细胞。使用太少或太多的细胞会导致消化过度或消化不足,这会降低文库的质量。此外,细胞的固定和剧烈的机械剪切趋于降低数据质量。
生物信息学数据分析由我们内部的生物信息学专家团队进行。标准的生物信息学分析包括:
序列分析:将测序读段比对到基因组,并删除重复的reads。
峰发现:使用MACS 2.1.0,将双端测序中的两个reads用于peak calling。
片段密度的确定:为了鉴定基因组的转座事件的密度,将基因组分为32 bp的条带,并确定每个条带中的片段数。为此,将reads扩展到200 bp以使数据平滑。通过随机采样对所有样本的比对reads数进行归一化,以使每一个样本包含所有样本中比对reads数最少样本相同数目的reads。
活动区域分析:为了比较两个或多个样本之间的峰,将重叠的峰分组为“活动区域”,这是由最上游峰的起始坐标和最下游峰的终止坐标定义的专有度量。