一、概述
CUT&RUN,全称为Cleavage Under Targets and Release Using Nuclease,由美国Fred Hutchinson癌症研究中心的Steven Henikoff团队开发。该技术的主要特点是,它可以在完整的细胞或细胞核上进行,无需甲醛固定或超声处理。细胞经过透化处理(扩孔)后与目标抗体孵育,抗体进入核内与目标蛋白结合;细胞再与蛋白A/G连接的MNase孵育,目标抗体Fc段结合Protein A-MNase或Protein A/G-MNase。然后激活MNase酶切割结合位点两边的染色质,这些经过切割的片段扩散到细胞核外,而未切割的部分留在核内,可大大降低背景。之后从上清液中收集DNA片段并测序。
CUT&Tag(Cleavage Under Targets and Tagmentation, CUT&Tag)是一种研究蛋白质与DNA互作的新方法。该方法通过分子生物学手段将高活性的Tn5转座酶与Protein A融合,并装载建库接头引物形成pA-Tn5转座复合物。在抗体的引导下该pA-Tn5转座复合物可靶向切割目的蛋白附近的DNA序列。与传统ChIP-Seq相比,该技术无需交联与超声打断操作,规避了其所引起的抗原决定簇遮盖和样本损失问题,提高了信噪比,并使所需细胞量减少(可低至60个)。与CUT&Run相比,该技术在pA-Tn5转座复合物进行切割时在切割片段的两端加上接头序列,可直接PCR建库,无需末端抹平和接头连接的操作,省时高效。CUT&Tag技术可从基因组范围内检测组蛋白、RNA polymerase II和转录因子等蛋白质结合的DNA区端,应用于临床和科研的表观遗传学研究。或是结合生物信息学分析,找到转录因子下游调控的靶基因,为进一步阐明生物学机制提供依据。
二、实验流程
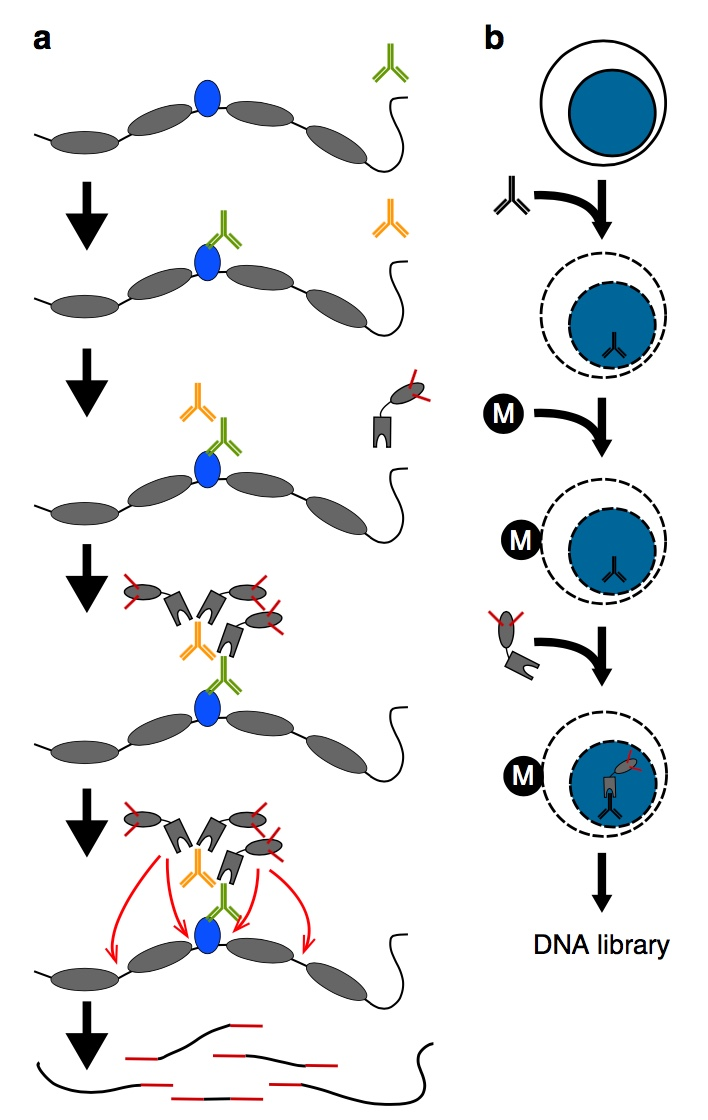
CUT&Tag
细胞先与包被刀豆蛋白A的磁珠(Concanavalin A-coated magnetic beads, ConA beads)吸附结合,方便后续操作。用非离子去污剂洋地黄皂苷(Digitonin)进行细胞穿孔后,依次孵育针对靶蛋白的一抗、相应的二抗、pA-Tn5融合蛋白。其中pA-Tn5中的pA可以识别二抗的Fc区域,Tn5酶在加入Mg2+后定向切割靶蛋白附近的DNA序列。且Tn5酶进行切割时会在切割片段的两端加上接头序列,因此切割产物可以直接PCR扩增建库,经纯化后用于高通量测序。
CUT&RUN
(1)未固定的核结合在lectin coated磁珠上;
(2)先后与抗体和蛋白A-MNase(pA-MN)孵育,其次是简单的洗涤;
(3)在冰上与Ca++混合,启动裂解反应,之后利用螯合作用停止数秒到数分钟;
(4)离心包含释放的TF-DNA复合物的上层清液,从而进行恢复。
(5)提取上清液中的DNA,建库测序。
上机测序
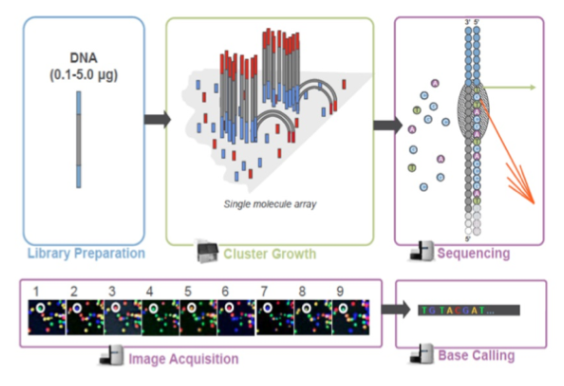
库检合格后,把不同文库按照有效浓度及目标下机数据量的需求 pooling 后进行 Illumina NovaSeq 测序。测序基于边合成边测序(Sequencing by Synthesis)的原理,在测序的 flow cell 中加入四种荧光标记的 dNTP、DNA 聚合酶以及接头引物进行扩增,在每一个测序簇延伸互补链时,每加入一个被荧光标记的 dNTP 就能释放出相对应荧光,测序仪通过捕获荧光信号,并通过计算机软件将光信号转化为测序峰,从而获得待测片段的序列信息。测序过程如下图所示:
生物信息分析流程
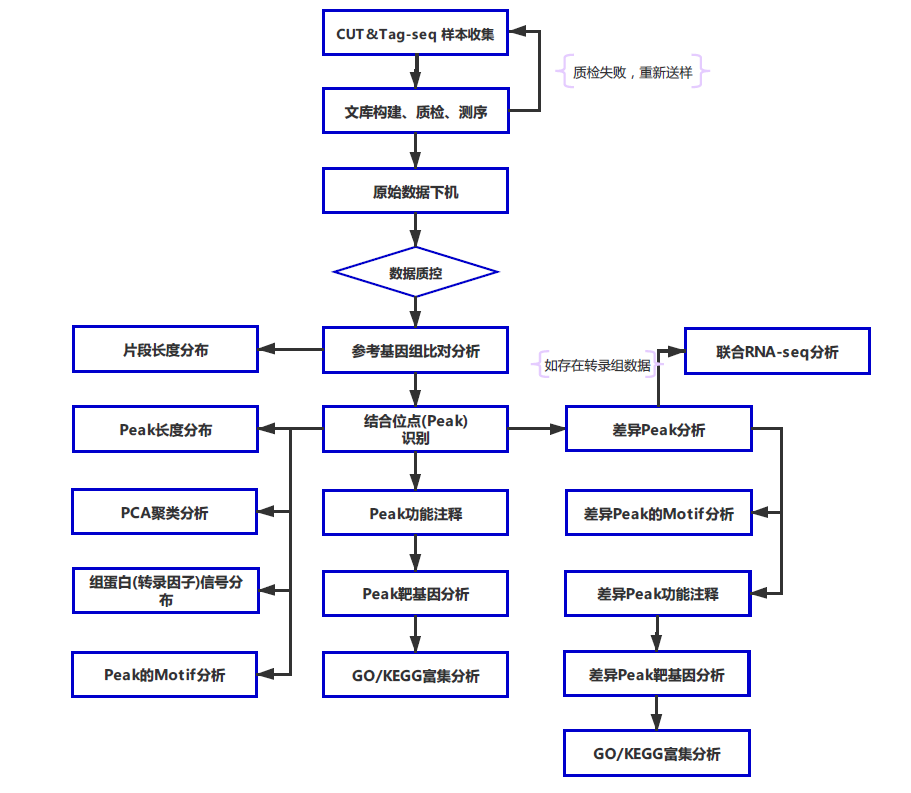
测序数据下机之后,获得原始测序序列(Raw data),在有相关物种参考序列或参考基因组的情况下,通过如下图标准流程进行生物信息分析:
三、应用范围
一般使用新鲜细胞、梯度冻存细胞、新鲜组织或是冰冻组织进行实验,可以广泛应用于哺乳动物的蛋白与DNA互作研究。酵母和植物等生物类型可经由破除细胞壁或者提取细胞核来进行实验。
四、技术优势
(1)实验材料无需交联,原位(In situ)操作;
(2)信噪比高,所需测序深度减少,性价比高;
(3)实验可重复性好;
(4)省时高效,从细胞收集到测序文库构建仅需1-2天。
五、结果展示
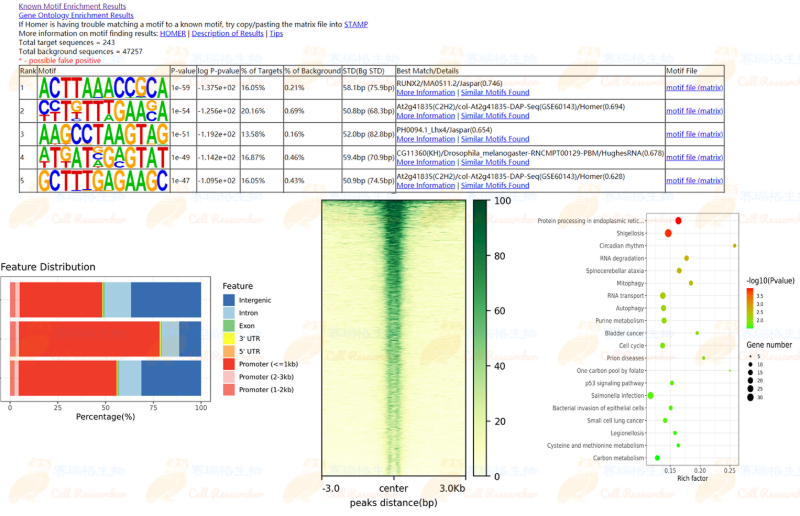
完整的PDF报告模板请联系销售获取。